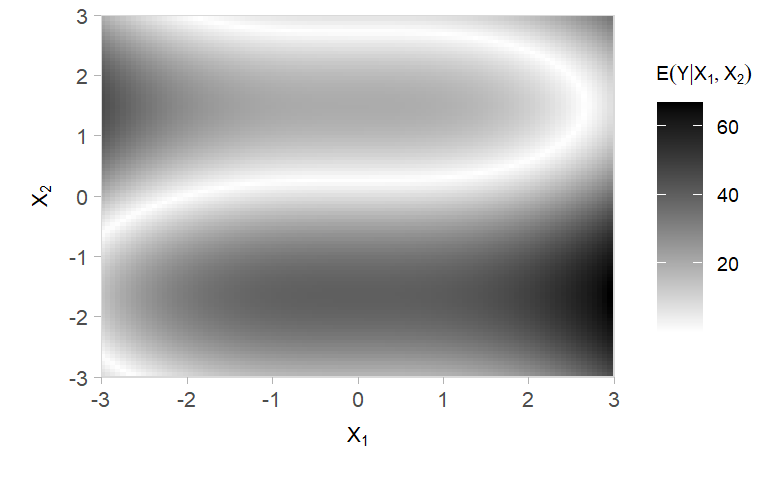
Use a regra da cadeia para encontrar expressões algébricas para o vetor gradiente \[
\nabla_{\boldsymbol{\phi}} J(\boldsymbol{\phi}) = \left(\frac{\partial J}{\partial w_1},
\ldots, \frac{\partial J}{\partial b_3} \right).
\]
Solução
Temos que \(\frac{\partial J}{\partial \hat{y}_i}\) será a derivada da função de custo, no caso MSE, definido por \(J = \frac{1}{m}\sum^m_{i=1}(\hat{y}-y)^2\). Desta forma, para cada \(i\), \(\frac{\partial J}{\partial \hat{y}_i} = 2(\hat{y}_i-y_i)\); e para o conjunto de todos as \(i\) observações, \(\frac{\partial J}{\partial \hat{y}} = \frac{2}{m}\sum_{i=1}^m(\hat{y}_i-y_i)\). Como isso é um escalar, chamarei \(\frac{\partial J}{\partial \hat{y}_i}\) de \(\epsilon_{1,i}\) (e, consequentemente, \(\frac{\partial J}{\partial \hat{y}}\) de \(\epsilon_{1} = \frac{1}{m}\sum_{i=1}^m\epsilon_{1,i}\)).
Para encontrar \(\nabla_{\boldsymbol{\Phi}}J(\boldsymbol{\Phi}) = (\nabla_{\Omega_1}J, \nabla_{\beta_1}J,\nabla_{\Omega_0}J,\nabla_{\beta_0}J)\), devemos aplicar a regra da cadeia, e retropropagar as derivadas parciais, da seguinte forma:
Como \(\hat{y}_i = h(f_{1,i})\); e \(f_{1,i} = \boldsymbol{\Omega}_1\boldsymbol{h}_{1,i}+\beta_1\); e \(\boldsymbol{h}_{1,i} = a(\boldsymbol{f}_{0,i})\); e \(\boldsymbol{f}_{0,i} = \boldsymbol{\Omega}_0\boldsymbol{x}_i + \boldsymbol{\beta}_0\), temos que:
\[
\frac{\partial J}{\partial f_{1,i}} = \frac{\partial J}{\partial \hat{y}_i} \frac{\partial \hat{y}_i}{\partial f_{1,i}} = \epsilon_{1,i} * h'(f_{1,i}) = \epsilon_{1,i} * 1 = \epsilon_{1,i} \text{ (Visto que h(.) é função identidade)}
\]
Aplicando a todas as \(i\) observações, teríamos que a perda total seria simplesmente
\[
\frac{\partial J}{\partial f_{1}} = \frac{1}{m}\sum_{i=1}^m\epsilon_{1,i} = \epsilon_1.
\]
Como \(\boldsymbol{f}_{1,i}=\boldsymbol{\Omega}_1\boldsymbol{h}_{1,i} + \beta_1\), calculamos
\[
\frac{\partial\boldsymbol{f}_{1,i}}{\partial\boldsymbol{\Omega}_1} = \boldsymbol{h}_{1,i}^T \text{ (Visto que } \nabla_{\boldsymbol{f}} = \boldsymbol{J_f}^T \text{)}, \text{ e}
\\
\frac{\partial\boldsymbol{f}_{1,i}}{\partial\beta_1} = 1.
\]
Com estes resultados, podemos calcular
\[
\frac{\partial J}{\partial \boldsymbol{\Omega}_1} = \frac{\partial J}{\partial f_{1,i}} \frac{\partial f_{1,i}}{\partial \boldsymbol{\Omega}_1} = \frac{1}{m}\sum_{i=1}^m\epsilon_{1,i} * \boldsymbol{h}_{1,i}^T \text{, e}
\]
\[
\frac{\partial J}{\partial \beta_1} = \frac{\partial J}{\partial \boldsymbol{f}_{1,i}} \frac{\partial \boldsymbol{f}_{1,i}}{\partial \beta_1} = \frac{1}{m}\sum_{i=1}^m\epsilon_{1,i} * 1.
\]
Agora, para obtermos \(\frac{\partial J}{\partial \boldsymbol{\Omega}_0}\) e \(\frac{\partial J}{\partial \boldsymbol{\beta}_0}\), basta seguir retropropagando as derivadas parciais utilizando a regra da cadeia.
Como \(\boldsymbol{f}_{1,i} = \boldsymbol{\Omega}_1\boldsymbol{h}_{1,i}+\beta_1\), e \(\boldsymbol{h}_{1,i}=a(\boldsymbol{f}_{0,i})\), temos que:
\[
\frac{\partial J}{\partial \boldsymbol{f}_{0,i}} = \frac{\partial J}{\partial \boldsymbol{h}_{1,i}} \odot a'(\boldsymbol{f}_{0,i}),
\]
onde \(a'(.)\) é a derivada da função sigmoide, escolhida como ativação para a camada oculta.
Derivando \(\frac{\partial \boldsymbol{f}_{1,i}}{\partial \boldsymbol{h}_{1,i}} = \nabla_{\boldsymbol{h}_{1,i}} (\boldsymbol{\Omega_1\boldsymbol{h}_{1,i}+\beta_1}) = \boldsymbol{\Omega}_1^T\). Então,
\[
\frac{\partial J}{\partial \boldsymbol{h}_{1,i}} = \frac{\partial J}{\partial \boldsymbol{f}_{1,i}} \frac{\partial \boldsymbol{f}_{1,i}}{\partial \boldsymbol{h}_{1,i}} = \epsilon_{1,i} * \boldsymbol{\Omega}_1^T,
\]
para cada \(i\). De maneira análoga,
\[
\frac{\partial J}{\partial \boldsymbol{h}_{1}} = \frac{1}{m} \sum^m_{i=1} \epsilon_{1,i} * \boldsymbol{\Omega}_1^T.
\]
E, desta forma,
\[
\frac{\partial J}{\partial \boldsymbol{f}_{0,i}} = (\epsilon_{1,i} * \boldsymbol{\Omega}_1^T) \odot a'(\boldsymbol{f}_{0,i}),
\]
para cada \(i\). Então,
\[
\frac{\partial J}{\partial \boldsymbol{f}_{0}} = \frac{1}{m} \sum_{i=1}^m (\epsilon_{1,i} * \boldsymbol{\Omega}_1^T) \odot a'(\boldsymbol{f}_{0,i}),
\]
será um vetor \(\nu_{(m_xp)}\). Desta forma, chamaremos \(\boldsymbol{\nu}_{0,i} = \frac{\partial J}{\partial \boldsymbol{f}_{0,i}} = (\epsilon_{1,i} * \boldsymbol{\Omega}_1^T) \odot a'(\boldsymbol{f}_{0,i})\), para cada \(i\).
Com estes resultados, podemos calcular diretamente os gradientes \(\frac{\partial J}{\partial \boldsymbol{\Omega}_0}\) e \(\frac{\partial J}{\partial \boldsymbol{\beta}_0}\), tal que:
\[
\frac{\partial J}{\partial \boldsymbol{\Omega}_0} = \frac{\partial J}{\partial \boldsymbol{f}_{0,i}} \frac{\partial \boldsymbol{f}_{0,i}}{\partial \boldsymbol{\Omega}_0} = \frac{1}{m}\sum_{i=1}^m\boldsymbol{\nu}_{0,i} * \boldsymbol{x}_i^T, \text{ e}
\]
\[
\frac{\partial J}{\partial \boldsymbol{\beta}_0} = \frac{\partial J}{\partial \boldsymbol{f}_{0,i}} \frac{\partial \boldsymbol{f}_{0,i}}{\partial \boldsymbol{\beta}_0} = \frac{1}{m}\sum_{i=1}^m\boldsymbol{\nu}_{0,i} * 1 = \frac{1}{m}\sum_{i=1}^m\boldsymbol{\nu}_{0,i}.
\]
Organizando então os resultados, temos
\[\begin{align*}
\nabla_{\phi}J(\phi) &= (\nabla_{\Omega_1}J, \nabla_{\beta_1}J,\nabla_{\Omega_0}J,\nabla_{\beta_0}J) \\
&= \left( \epsilon_{1} \boldsymbol{h}_{1}^T, \epsilon_1, \boldsymbol{\nu}_0 \mathbf{x}^T, \boldsymbol{\nu}_0 \right).
\end{align*}\]